Version complète: [Taxonomie] - Phylogénie des Formicidae
Acideformik.com > AcideFormik :: Myrmécologie > Informations sur la biologie/comportement des fourmis
voici le lien ou on peut voir l'arborescence de cette phylogénie
 http://tolweb.org/tree?group=Formicidae&am...group=Vespoidea
http://tolweb.org/tree?group=Formicidae&am...group=Vespoidea
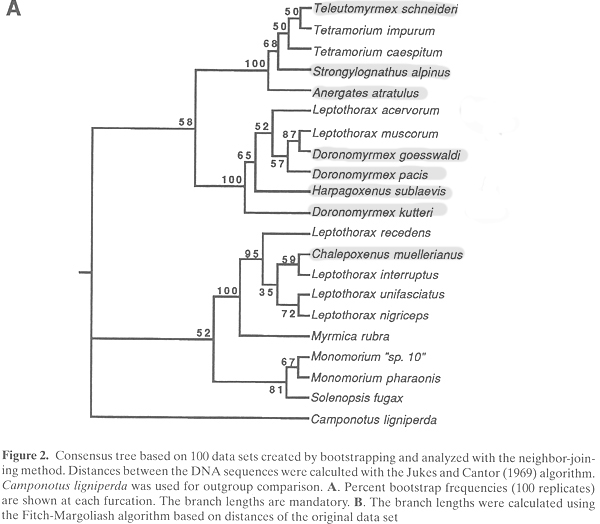
développement qui doit dater car y figure des Leptothorax
C'est l'objet d'une conversation sur un forum américain.
De ce que j'ai pu comprendre, tous les scientifiques ne sont pas d'accord. La référence serait Bolton mais les travaux sont en cours. L'une des difficulté serait la difficulté de trouver des fossiles pour retracer l'évolution. On en trouve dans l'ambre, rarement.
c'est toujours le meme problème avec la phylogénie
tout dépend du matériel qu'on utilise pour construire un arbre
le DNA ici , le choix de la séquence mais on peut se baser sur un paquet de séquences différentes au sein du DNA comme les ITS par exemple,etc...
tu sais sur quelle région ils se sont basés?
et puis il n'y a pas que le DNA que l'on peut utiliser.....
Pour l'arbre qu'ils ont sorti
je pense qu'ils ont utilisé la parcimonie
et tout dépend de la technique
en ce moment c'est l'AFLP qui a le vent en poupe
apres le matériel vient le choix de la méthode utilisée pour traiter les données (maximum de parcimonie, maximum de vraisemblance, inférence bayésienne, etc....)
et la encore ca peut fluctuer
bref c'est souvent difficile d'affirmer quelque chose et il faut analyser ce qu'on cherche à montrer pour faire le meilleur choix du matériel et de la méthode
quand on regarde cet arbre
il y a des choses qui sont déja pas terrible dès le départ
52% pour le bootstrap en ce qui concerne le groupe monophylétique du bas
la valeur de 35 n'est pas géniale non plus
c'est issu de quel article? (si tu as les renseignements biensûr) :wink:
tout dépend du matériel qu'on utilise pour construire un arbre
le DNA ici , le choix de la séquence mais on peut se baser sur un paquet de séquences différentes au sein du DNA comme les ITS par exemple,etc...
tu sais sur quelle région ils se sont basés?
et puis il n'y a pas que le DNA que l'on peut utiliser.....
Pour l'arbre qu'ils ont sorti
je pense qu'ils ont utilisé la parcimonie
et tout dépend de la technique
en ce moment c'est l'AFLP qui a le vent en poupe
apres le matériel vient le choix de la méthode utilisée pour traiter les données (maximum de parcimonie, maximum de vraisemblance, inférence bayésienne, etc....)
et la encore ca peut fluctuer
bref c'est souvent difficile d'affirmer quelque chose et il faut analyser ce qu'on cherche à montrer pour faire le meilleur choix du matériel et de la méthode
quand on regarde cet arbre
il y a des choses qui sont déja pas terrible dès le départ
52% pour le bootstrap en ce qui concerne le groupe monophylétique du bas
la valeur de 35 n'est pas géniale non plus
c'est issu de quel article? (si tu as les renseignements biensûr) :wink:
un article en anglais .pdf
intéressant sur ce sujet
 Phylogeny of ants (Formicidae) based on morphology and DNA sequence data
Phylogeny of ants (Formicidae) based on morphology and DNA sequence data
intéressant sur ce sujet
Le moins que j'ai pu comprendre, c'est que c'est contreversé.
Il y aurait trois groupes :
- Poneroid : Ponerinae, Cerapachyinae, Leptanillinae et fourmis d'armée
- Formicoid : Dolichoderinae, Formicinae
- Myrmecoid : Myrmicinae, Myrmeciinae, Pseudomyrmecinae, Nothomyrmeciinae
Trois sous familles (Dolichoderinae, Formicinae et Pseudomyrmecinae) apparaissent toujours comme monophyletiques. Pas de noeud, elle ressemblent à ce qu'elles étaient il y a des millions d'années.
Le progrès viendra d'une meilleure connaissance de la structure interne du Ponerinae et du Myrmicinae. Et que l'ambre permet d'étudier les fossiles, rares, de leurs ancêtres.
lien sur l'ambre
Il y aurait trois groupes :
- Poneroid : Ponerinae, Cerapachyinae, Leptanillinae et fourmis d'armée
- Formicoid : Dolichoderinae, Formicinae
- Myrmecoid : Myrmicinae, Myrmeciinae, Pseudomyrmecinae, Nothomyrmeciinae
Trois sous familles (Dolichoderinae, Formicinae et Pseudomyrmecinae) apparaissent toujours comme monophyletiques. Pas de noeud, elle ressemblent à ce qu'elles étaient il y a des millions d'années.
Le progrès viendra d'une meilleure connaissance de la structure interne du Ponerinae et du Myrmicinae. Et que l'ambre permet d'étudier les fossiles, rares, de leurs ancêtres.
lien sur l'ambre
CITATION(k2rantitache)
tu sais sur quelle région ils se sont basés?
........
c'est issu de quel article? (si tu as les renseignements biensûr) :wink:
........
c'est issu de quel article? (si tu as les renseignements biensûr) :wink:
Here I post a tree, one of a couple more, based on genetic data, all showing the social parasites of Temnothorax (sensu Bolton) and of Leptothorax (sensu Bolton) on separate branches, together with the respective hosts (same for the parasites of Tetramorium). This tree is from Baur et al. 1996, Ins. Soc. 43, 53-67. (The grouping of Temnothorax with Myrmica and Monomorium, and of Leptothorax with Tetramorium in this instance is not well supported and pro
cékiki : bizzare, En général l'étude des fossiles n'est pas d'une grande utilité pour des arbres construits avec de telles methodes moléculaires. (à moins de vouloir récupérer de l'ADN dans les fossiles prisioniers de l'Ambre... OUAAAHH c'est jurassic Park...  Ceci dit c'est peut être faisable )
Ceci dit c'est peut être faisable )
(k2r : Arf "bootstrap" ça me rappelle de lointains souvenirs. c'est vrai que des valeurs de 50 .. boaf. Tiens la position de l'outgroup est étrange... (la branche de l'outgroup devrait pas plutot partir de avant le noeud commun ? ) )
Il est fort possible aussi que les phylogénies moléculaires tournent en rond pendant un moment au niveau des sous familles... tout dépends des crédits que l'on allouera pour les séquençages. ensuite il faut parfois de longues années pour avoir des arbres congruants (qui racontent la même chose)... l'idéal est de faire un petit milliers de phylogénies avec des molécules diférentes et indépendantes. je doute que chez les fourmis on ait séquencé autant de gènes ... En plus vu la structure génétique particulières de certaines population je ne suis pas sûr que l'on ne se réserve pas de petites surprises si on veut faire la phylogénie des groupes inférieurs aux sous familles...
Ainsi depuis trente ans les classifications bougent bougent bougent dans à peu près tous les groupes ... visiblement les fourmis ne font pas exception.
(k2r : Arf "bootstrap" ça me rappelle de lointains souvenirs. c'est vrai que des valeurs de 50 .. boaf. Tiens la position de l'outgroup est étrange... (la branche de l'outgroup devrait pas plutot partir de avant le noeud commun ? ) )
Il est fort possible aussi que les phylogénies moléculaires tournent en rond pendant un moment au niveau des sous familles... tout dépends des crédits que l'on allouera pour les séquençages. ensuite il faut parfois de longues années pour avoir des arbres congruants (qui racontent la même chose)... l'idéal est de faire un petit milliers de phylogénies avec des molécules diférentes et indépendantes. je doute que chez les fourmis on ait séquencé autant de gènes ... En plus vu la structure génétique particulières de certaines population je ne suis pas sûr que l'on ne se réserve pas de petites surprises si on veut faire la phylogénie des groupes inférieurs aux sous familles...
Ainsi depuis trente ans les classifications bougent bougent bougent dans à peu près tous les groupes ... visiblement les fourmis ne font pas exception.
CITATION(Elrou)
cékiki : bizzare, En général l'étude des fossiles n'est pas d'une grande utilité pour des arbres construits avec de telles methodes moléculaires. (à moins de vouloir récupérer de l'ADN dans les fossiles prisioniers de l'Ambre... OUAAAHH c'est jurassic Park...  Ceci dit c'est peut être faisable )
Ceci dit c'est peut être faisable )
Les disciplines qui contribuent à la phylogénie sont effectivement la phylogénie moléculaire, la zoologie, l'embriologie, la caryologie ( structure des chromosomes ?) , l'anatomie comparée ...et la paléontologie.
Ce n'est pas une preuve mais la personne concernée dont je tiens le propos est crébible ( chercheur au CNRS sur Perpignan, labo concerné par le partenariat) : A Tautavel( 66) en recueille les sédiments à bien des fins. Ils rechercheraient la microfaune fossilisée.
Hem en générale la microfaune est effectivmeent receuillies sur les sites paléontologiques... mais dans ces disciplines on entend le plus souvent par microfaune les micromamifères (campagnoles etc). Cela sert à reconstituer les paléoclimats et la végétation.
Juste je voulais dire que l'arbre plus haut est un phenogramme avec des données moléculaires. on fait qussi des cladogrammes avec des donnes morphologiques (obtenues chez les fossiles) mais chez les insectes je ne suis pas au courant. ce doit être faisable.
Pour les fossiles dans l'ambre ma blague sur Jurassik Park n'en est pas vraiment une je pense que certaines équipes doivent avoir l'espoir de récupérer quelques gènes dans des fossiles prisonniers afin de les faire rentrer dans un arbres construits avec des methodes moléculaires. En ce sens tu as raisons. la solution des arbres sur les espèces actuelles passent peut être par l'étude des gènes fossiles... Si c'est ça, c'est vertigineux je trouve.
Juste je voulais dire que l'arbre plus haut est un phenogramme avec des données moléculaires. on fait qussi des cladogrammes avec des donnes morphologiques (obtenues chez les fossiles) mais chez les insectes je ne suis pas au courant. ce doit être faisable.
Pour les fossiles dans l'ambre ma blague sur Jurassik Park n'en est pas vraiment une je pense que certaines équipes doivent avoir l'espoir de récupérer quelques gènes dans des fossiles prisonniers afin de les faire rentrer dans un arbres construits avec des methodes moléculaires. En ce sens tu as raisons. la solution des arbres sur les espèces actuelles passent peut être par l'étude des gènes fossiles... Si c'est ça, c'est vertigineux je trouve.
Abus de langage : j'aurai du employer le terme mésofaune.
CITATION
l'outgroup est étrange... (la branche de l'outgroup devrait pas plutot partir de avant le noeud commun ? )
Pas forcément cela dépend de la méthode utilisée pour construire l'arbre en fait.
Le plus intéressant est de faire des analyses sur des données morpho et moléculaires pour comparer et d'utiliser différentes méthodes et différentes régions conservées.
Ce sont surtout au niveau des données moléculaires que ca peut se compliquer en fonction du matériel et de sa région que l'on choisit
parfois on peut obtenir des choses complètement contradictoire....
Tout à fait d'accord avec toi k2r mais quelquesoit la methode je vois pas bien où est l'ancêtre commun de l'outgroup et du groupe dans l'arbre de cékiki. le noeud le plus à gauche est double (ce n'est pas un noeud dichotomique en fait). c'est pas très orthodoxe mais je pense que c'est un abus... (il ne devrait pas y avoir de branche verticale entre l'out groupe et le groupe... enfin bref c'est un détail).
Sinon si je me souviens bien, on fait habituellement plusieurs arbres, et on retiens l'arbre "majoritaire". mais pour faire cela de façon statistique il faut des arbres construits à partir de pleins de régions... et le séquençage chez la fourmis je ne sais pas si ça intérresse beaucoup de monde...
Sinon c'est chouette quand les methodes basées sur la cladistique (les caractères morphologiques) sont en accord avec les arbres moléculaires. MAis la cladistique chez les insectes ça doit être assez limité vu l'absence relative de fossiles anciens.
Il faut donc sans doute attendre pour caler les arbres ... (maisbon je ne sais pas peut être que la classif des fourmis possèdent des intérêts économiques majeurs que je ne connais pas et que des tas de labo bossent dessus ? ?)
?)
Pour cékiki : l'abus de langage c'est pas toi qui le fait ce sont les paléontologues. quand on passe son temps à déterrer des mamouths, les campagnoles c'est petit (=> microfaune est effectivement utilisé pour les campagnoles...) et les fourmis ... ben pour eux ça n'existe pas .
.
Sinon si je me souviens bien, on fait habituellement plusieurs arbres, et on retiens l'arbre "majoritaire". mais pour faire cela de façon statistique il faut des arbres construits à partir de pleins de régions... et le séquençage chez la fourmis je ne sais pas si ça intérresse beaucoup de monde...
Sinon c'est chouette quand les methodes basées sur la cladistique (les caractères morphologiques) sont en accord avec les arbres moléculaires. MAis la cladistique chez les insectes ça doit être assez limité vu l'absence relative de fossiles anciens.
Il faut donc sans doute attendre pour caler les arbres ... (maisbon je ne sais pas peut être que la classif des fourmis possèdent des intérêts économiques majeurs que je ne connais pas et que des tas de labo bossent dessus ?
Pour cékiki : l'abus de langage c'est pas toi qui le fait ce sont les paléontologues. quand on passe son temps à déterrer des mamouths, les campagnoles c'est petit (=> microfaune est effectivement utilisé pour les campagnoles...) et les fourmis ... ben pour eux ça n'existe pas
Ouaip ils n'ont surement pas d'ancêtre commun ou la branche alors devait etre trop longue (je pense que ca doit etre pour ca plutot)
Ce qui devait les intéresser c'était juste d'avoir leur outgroup et puis c'est tout;=)
Ca dépend de la méthode mais tu dois arriver à un arbre consensus.
Je crois qu'il y a actuellement pas mal de séquencage au niveau des fourmis du fait des nouvelles techniques et nouveaux appareils
suffit de regarder les données disponibles sur ncbi
:wink:

Ce qui devait les intéresser c'était juste d'avoir leur outgroup et puis c'est tout;=)
CITATION
Sinon si je me souviens bien, on fait habituellement plusieurs arbres, et on retiens l'arbre "majoritaire"
Ca dépend de la méthode mais tu dois arriver à un arbre consensus.
Je crois qu'il y a actuellement pas mal de séquencage au niveau des fourmis du fait des nouvelles techniques et nouveaux appareils
suffit de regarder les données disponibles sur ncbi
:wink:
CITATION
maisbon je ne sais pas peut être que la classif des fourmis possèdent des intérêts économiques majeurs que je ne connais pas et que des tas de labo bossent dessus ?
C'est vrai mais vu le nombre d'espèces de fourmis ... je crois qu'on peut attendre longtemps avant des phylogénies solides et complètes.
MAis tu as raison les technique de séquençage automatique et d'amplification d'ADN ne cessent de s'améliorer. Aujourd'hui une seule personne peut assurer le séquençage de plusieurs millier de paires de bases par jour (et encore je sous estime peut être ça fait un moment que je n 'ai pas mis les pieds dans un labo ni même que je n'ai croisé un chercheur (<= espèce en voie de disparition ?).
Un truc rigolo : dans les labos que je visitais il y a pas mal d'années le problème n'était pas de séquencer mais d'identifier les bestioles : des labos capables de séquencer des centaines des milliers de paires de bases y'en a plein en France. Mais trouver un spécialiste des hyménoptères pour certaines familles, par exemple, c'est coton. D'où des problèmes pour savoir les gènes de quoi on séquence . Fou , non ?
. Fou , non ?
Au rythme où on favorise les disciplines naturalistes en France j'ai bien peur qu'on ne finisse par faire de la phylogénie que pour ratus Norvegicus. Et encore y'en a qui confondent avec la souris .
.
MAis tu as raison les technique de séquençage automatique et d'amplification d'ADN ne cessent de s'améliorer. Aujourd'hui une seule personne peut assurer le séquençage de plusieurs millier de paires de bases par jour (et encore je sous estime peut être ça fait un moment que je n 'ai pas mis les pieds dans un labo ni même que je n'ai croisé un chercheur (<= espèce en voie de disparition ?).
Un truc rigolo : dans les labos que je visitais il y a pas mal d'années le problème n'était pas de séquencer mais d'identifier les bestioles : des labos capables de séquencer des centaines des milliers de paires de bases y'en a plein en France. Mais trouver un spécialiste des hyménoptères pour certaines familles, par exemple, c'est coton. D'où des problèmes pour savoir les gènes de quoi on séquence
Au rythme où on favorise les disciplines naturalistes en France j'ai bien peur qu'on ne finisse par faire de la phylogénie que pour ratus Norvegicus. Et encore y'en a qui confondent avec la souris
Ceci est une version "bas débit" de notre forum. Pour voir la version complète avec plus d'information, la mise en page et les images, veuillez cliquer ici.